Análisis de interacciones en el docking molecular
La semana pasada aprendimos a llevar a cabo docking macromolecular entre la proteína p53 y MDM2. En la semana de hoy vamos a llevar a cabo un estudio previo al del docking molecular, vamos a analizar la interacción entre la proteína MDM2 y el ligando Benzodiacepina.
¿Qué herramientas se van a emplear?
PDBSum. Base de datos pública que aporta información generalizada de los contenidos de cada una de las estructuras 3D depositadas en Protein Data Bank (PDB). Muestra las moléculas que constituyen la interacción además de diagramas esquemáticos de las interacciones. (http://www.ebi.ac.uk/thornton-srv/databases/cgi-bin/pdbsum/GetPage.pl?pdbcode=index.html)
LIGPLOT. Su uso para la entrada de hoy es opcional. Requiere una licencia académica para poder descargarse. La descarga es sencilla e intuitiva. Programa que genera esquemas de las interacciones ligando-proteína para un archivo de pdb dado. Las interacciones mostradas son aquellas mediadas por puentes de hidrógeno e interacciones hidrofóbicas. (https://www.ebi.ac.uk/thornton-srv/software/LigPlus/download.html)
Binding Database. Base de datos pública de las afinidades de interacción, enfocada en las interacciones de proteínas consideradas como potenciales dianas para fármacos. (https://www.bindingdb.org/bind/index.jsp)
¿Por qué es importante analizar las interacciones?
Para entender el efecto que pueda ejercer un compuesto sobre una proteína determinada es importante caracterizar la interacción que se establece entre ambas. A la hora de diseñar un fármaco parámetros como la constante de disociación (KD) o la concentración media inhibitoria (IC50) son cruciales.
Además, de cara a futuras entradas nos resultará importante extraer información acerca de la interacción proteína ligando. Para poder analizar los resultados de re-docking proteína-ligando hay que conocer qué interacciones se establecen.
¿Qué interacción vamos a estudiar?
En la semana de hoy vamos a seguir estudiando la proteína MDM2 pero esta vez se analizará su interacción con la Benzodiacepina.
La proteína MDM2 (de sus siglas en inglés “mouse double minute 2”) es una proteína crítica en la regulación negativa de la proteína supresora de tumores p53. Es un oncogen que inhibe la función de p53 induciendo la aparición de tumores. Se consolidó su concepción como oncogen en un estudio que determinó la sobre-expresión de este gen en 1 de cada 3 sarcomas (Oliner et al. 1992). Hay 3 mecanismos por los que MDM2 interfiere en las funciones de p53: (1) inhibición al interaccionar con p53 “tapando” su dominio de activación (2) al marcar p53 para degradación (3) al exportar p53 del núcleo al citoplasma.
Se han estudiado por tanto el uso de inhibidores de la interacción MDM2-p53 para ejercer un efecto anti-tumoral. Johnson & Johnson reportaron una clase de benzodiazepina (BDP), 1,4-benzodiacepina-2,5-diona una molécula de pequeño tamaño, como inhibidor de la interacción con p53 (PDB ID 1T4E).
Tutorial del Análisis de Interacciones
- En primer entraremos en el servidor web PDBSum donde buscaremos nuestro código PDB (1T4E). Nos encontraremos con una página como la siguiente:

- Tras llevar a cabo la búsqueda nos aparecerá una página con 7 pestañas. En la primera “Top Page” nos aparecerá información general que concierne las estructuras contenidas en el archivo PDB, la reacción enzimática que se produce y el artículo científico que publicó la estructura. En la parte de la izquierda viene representada la estructura, vemos que se representan dos péptidos interaccionando con dos ligandos.
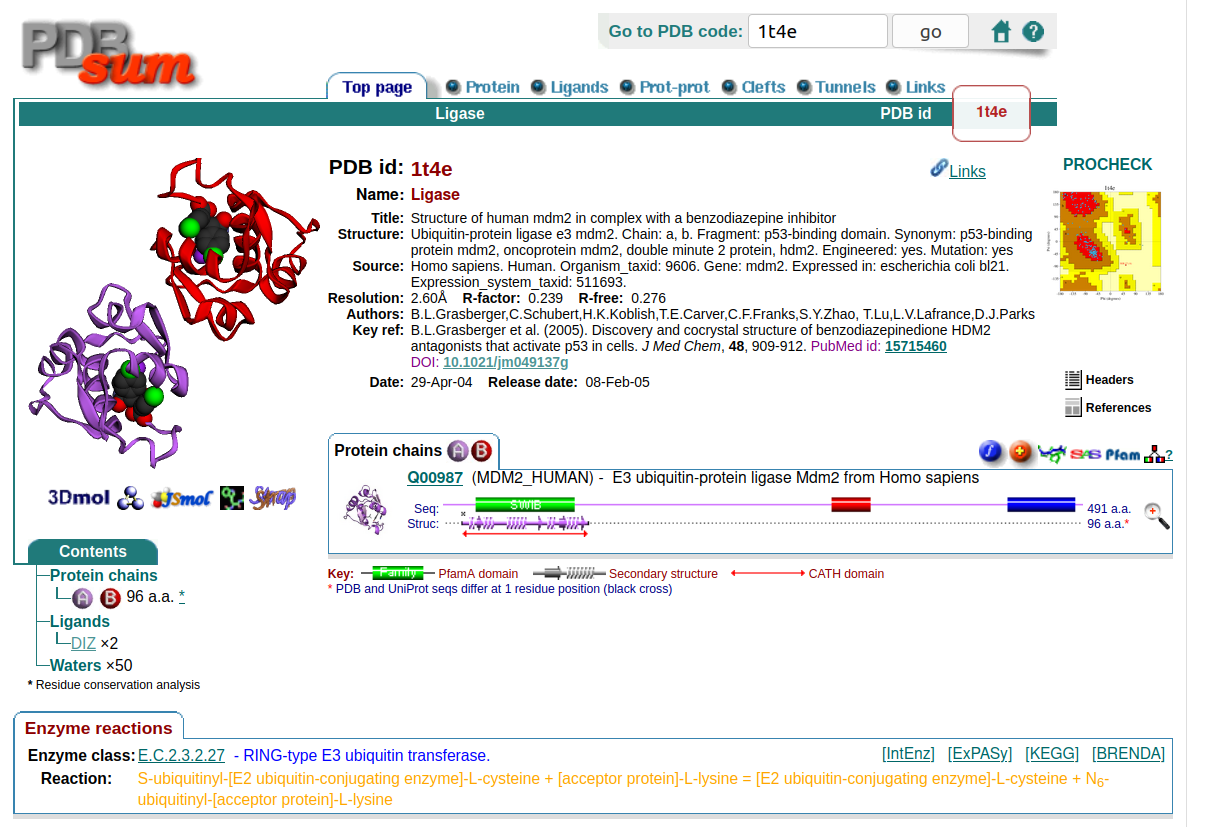
- Este archivo PDB contiene un homodímero de MDM2 con los péptidos de cada monómero que interaccionan con la benzodiacepina. Estos péptidos reciben el nombre de cadena A y cadena B. Para comprobar que efectivamente se trata de dos cadenas idénticas, le damos a la pestaña de “Protein” donde efectivamente nos indica que “Protein chain A representing identical chain: B” junto con la secuencia y estructura secundaria debajo.
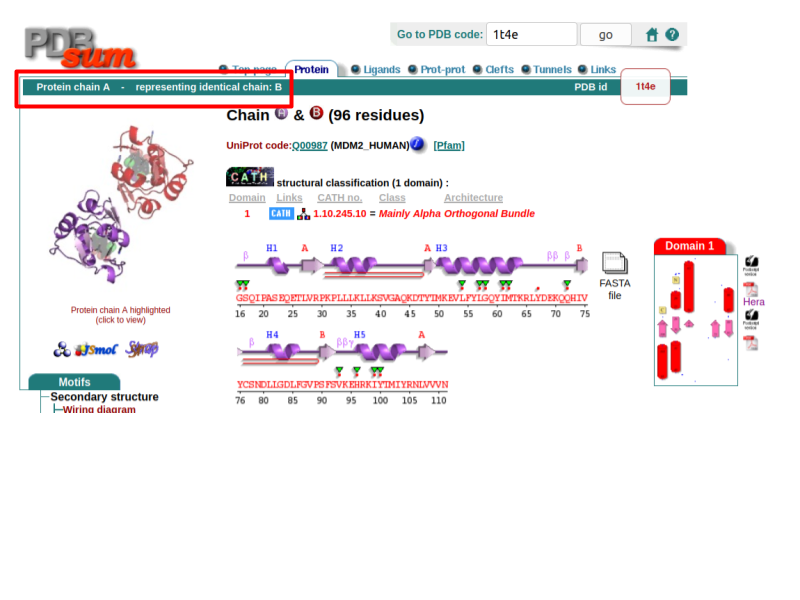
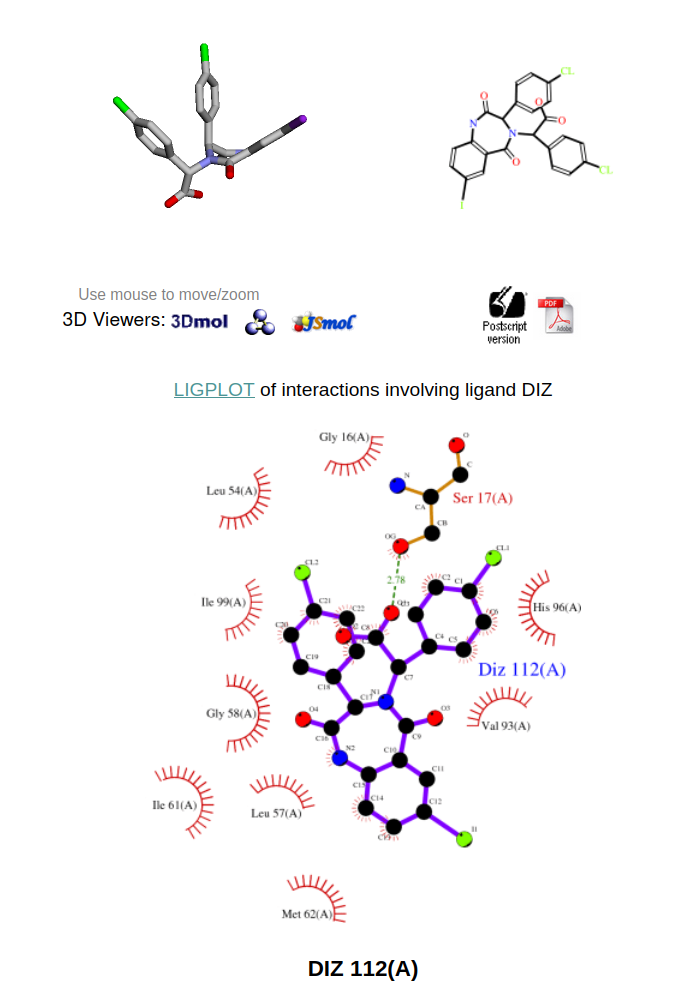
- Nos interesa de momento extraer información acerca del ligando. Por tanto nos iremos a la pestaña de “Ligands” donde nos vendrá información acerca de la estructura del ligando y su interacción. Nos representa la interacción entre los residuos de MDM2 y la Benzodiacepina para la cadena A y nos indica que las interacciones son similares para la cadena B. Para el propósito de la entrada de hoy y la semana que viene se analizarán las interacciones de la cadena A. Los residuos rodeados en semi círculos rojos indican interacciones hidrofóbicas mientras que la línea intermitente verde indica un puente de hidrógeno. Se recoge en la Tabla Suplementaria 1 al final de esta entrada el listado de estas interacciones.
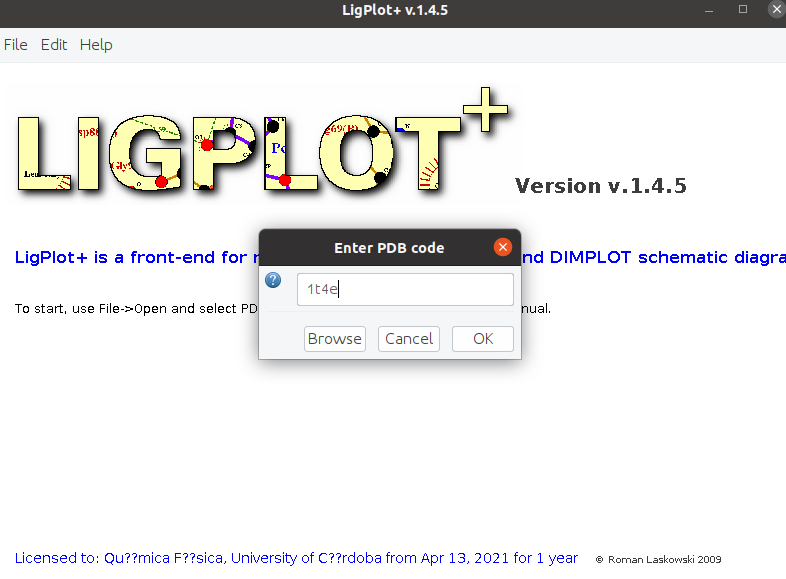
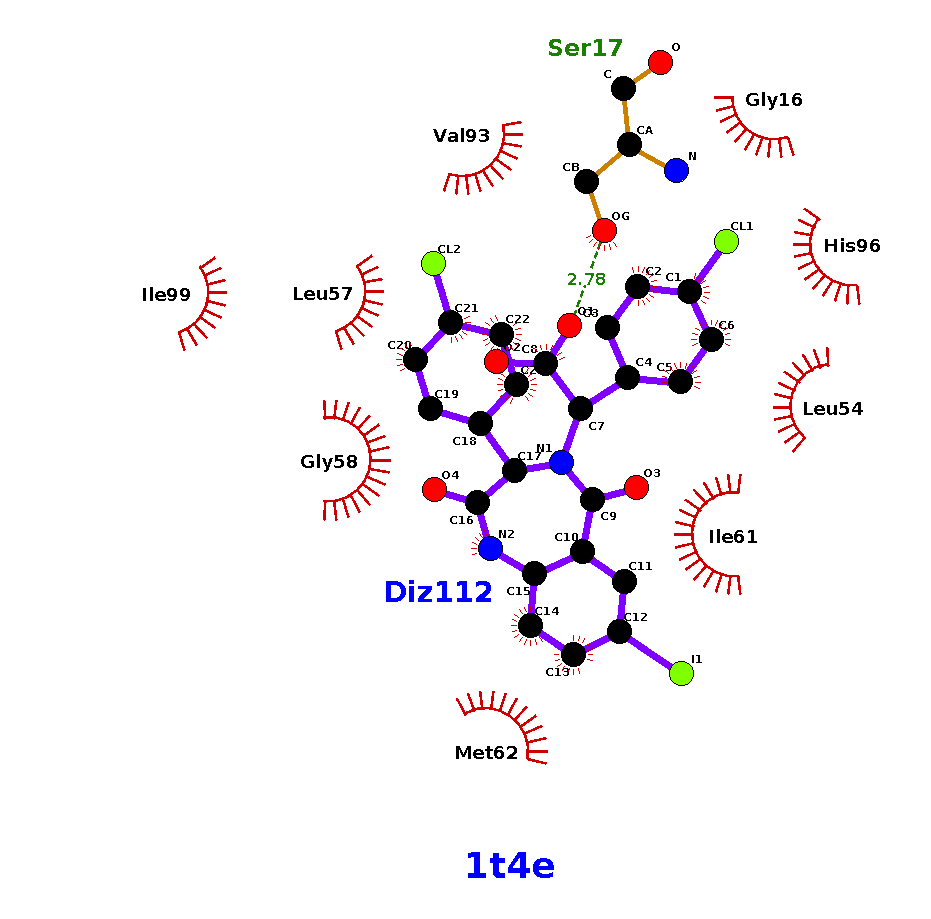
- Alternativamente podríamos emplear el programa LIGPLOT+ para obtener el mismo tipo de información. No es necesario para la entrada de hoy. De todas formas se hará un breve resumen de como correr este programa. Tras descargar el programa (instalación intuitiva sin complicación) abriremos la pestaña de File y le daremos a la opción de “Open PDB File”. Buscaremos el nombre de nuestro archivo PDB (no tiene por qué estar descargado) y nos abrirá una ventana con los ligandos que interaccionan con la proteína en el archivo PDB. Seleccionamos en nuestro caso la cadena A, y obtendremos una figura muy similar y con las mismas interacciones que en el apartado 4.
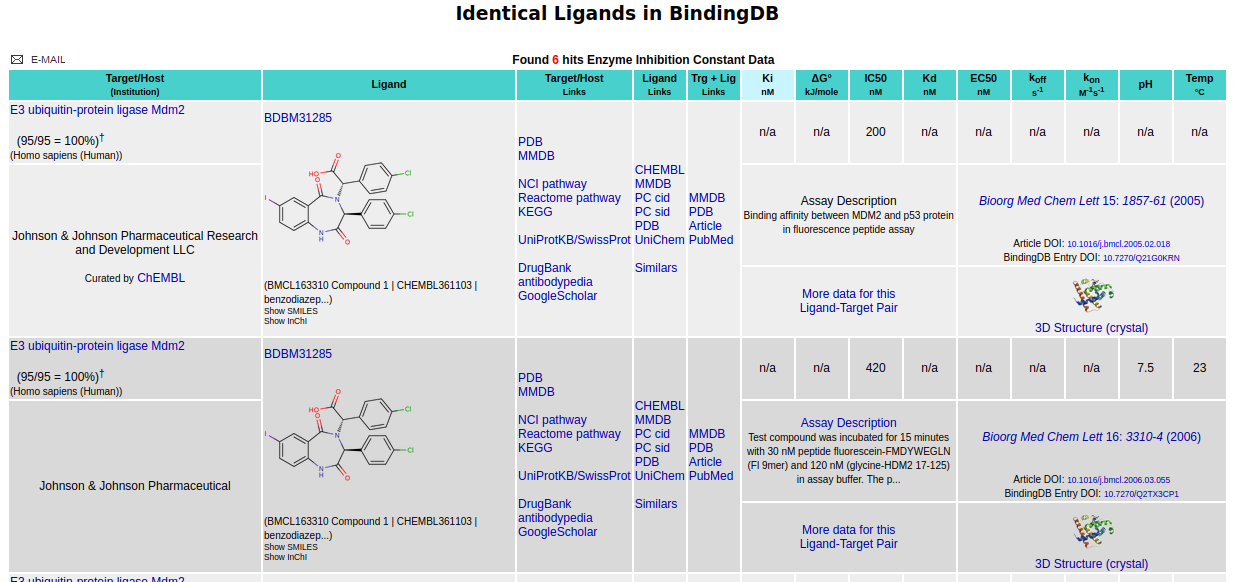
- Por último vamos a llevar a cabo una búsqueda de la estrucutra PDB en el servidor Binding Database para poder valorar la afinidad de unión entre nuestra proteína y ligando.

- El resultado de la búsqueda es una tabla donde nos indica la publicación científica, el ligando, y datos termodinámicos de la interacción. Como resultados relevantes tenemos que según Cummings et al. la constante de disociación (KD) tiene un valor de 80 nM mientras que Wells & McClendon determinaron el valor de la constante en 67 nM. Se ha descrito en dos estudios diferentes el valor de IC50 como 420 nM y en otro estudio como 200 nM (M. Kemp, Weïwer & N.Koehler 2012)(Wells & McClendon 2007)(Marugan & et al. 2005).
Conclusión
Hoy hemos aprendido a caracterizar las diferentes interacciones que se establecen entre un ligando y una proteína para un archivo PDB determinado. Hemos determinado los residuos que interaccionan, la naturaleza de la interacción y parámetros termodinámicos del complejo proteína-ligando.
Lo próximo
Se seguirá indagando en el campo de docking por medio de diferentes estrategias y recursos.
Bibliografía
| Tabla S1. Listado de AAs De MDM2 que interaccionan en el complejo MDM2 Cadena A- Benzodiacepina | |
| Aminoácido | Tipo de Interacción |
| Ser17 | Puente de Hidrógeno |
| Gly16 | Interacción Hidrofóbica |
| Val93 | Interacción Hidrofóbica |
| His96 | Interacción Hidrofóbica |
| Leu57 | Interacción Hidrofóbica |
| Leu54 | Interacción Hidrofóbica |
| Ile99 | Interacción Hidrofóbica |
| Gly58 | Interacción Hidrofóbica |
| Ile61 | Interacción Hidrofóbica |
| Met62 | Interacción Hidrofóbica |