Docking Macromolecular
Docking Macromolecular
La semana pasada se realizó Blind Docking Proteína-Ligando la interacción de la proteína Cdc25B con el ligando menadiona. En cambio, esta semana vamos a aprender a hacer Docking Macromolecular.
¿Qué herramientas se van a emplear?
HDOCK (http://hdock.phys.hust.edu.cn/), un servidor web gratuito que permite realizar docking proteína-proteína así como proteína-ADN/ARN.
UFC Chimera, programa tratado en otras entradas para visualizar las estructuras macromoleculares.
¿Por qué es importante el Docking Macromolecular?
Las interacciones que se establecen entre macromoléculas son cruciales para multitud de procesos biológicos como la transducción de señales, la regulación celular, la síntesis de proteínas, la replicación y reparación de ADN, la transcripción de ARN etc. Determinar las interacciones macromoleculares permite entender el mecanismo de procesos biológicos donde están involucrados estos complejos y descubrir nuevos fármacos que mejoren o dificulten dicha asociación. Realizar docking permite predecir estas interacciones de forma computacional, ahorrando tanto los altos costes como las dificultades técnicas de estudiar estas interacciones por métodos experimentales.
¿Qué interacción vamos a estudiar?
Se va a estudiar la interacción entre la proteína MDM2 y p53.
El gen p53 es un factor de transcripción bien descrito, que juega un papel crucial en la regulación del ciclo celular, la reparación de ADN, la diferenciación celular y apoptosis. p53 actúa en respuesta a daño celular induciendo la detención del ciclo celular y apoptosis. La inactivación de p53 es una causa común de transformaciones neoplásicas.
La proteína MDM2 (de sus siglas en inglés “mouse double minute 2”) es una proteína crítica en la regulación negativa de la proteína supresora de tumores p53. Es un oncogen que inhibe la función de p53 induciendo la aparición de tumores. Se consolidó su concepción como oncogen en un estudio que determinó la sobre-expresión de este gen en 1 de cada 3 sarcomas (Oliner et al. 1992). Hay 3 mecanismos por los que MDM2 interfiere en las funciones de p53: (1) inhibición al interaccionar con p53 “tapando” su dominio de activación (2) al marcar p53 para degradación (3) al exportar p53 del núcleo al citoplasma.
Tutorial de Docking Macromolecular
- En primer entraremos en el servidor web HDCOK (http://hdock.phys.hust.edu.cn/). Nos encontraremos con una página como la siguiente:
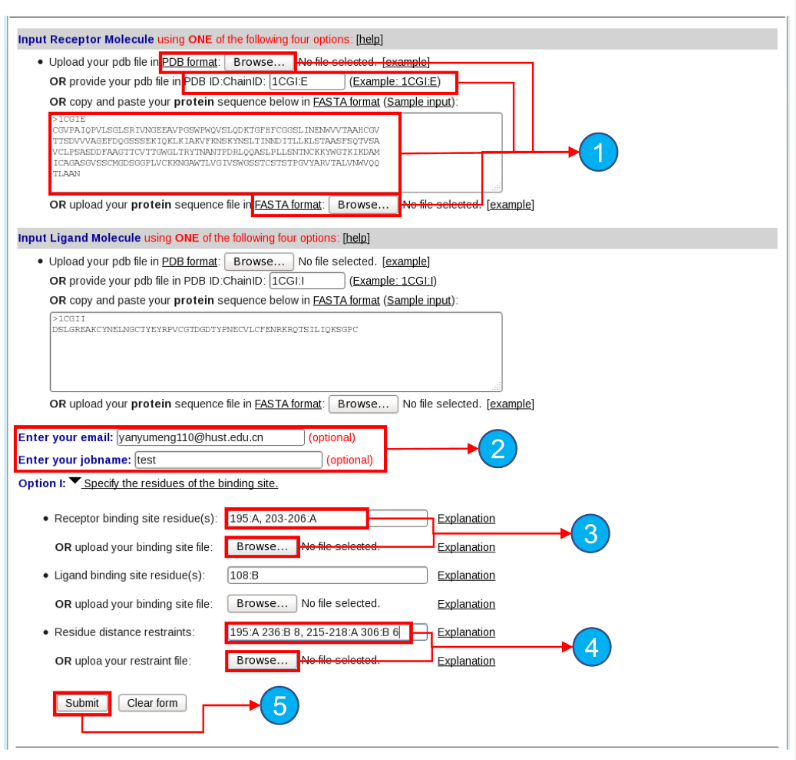
Aparecen varias opciones. Vamos a comentar cada una de ellas: (1) Muestra las diferentes opciones para introducir la macromolécula – se puede subir la macromolécula descargada en formato PDB, introducir el identificador PDB de la proteína pudiendo definirla cadena en el caso que proceda o introducir el código FASTA (2) Permite introducir nuestro correo para que el servidor nos avise cuando los resultados estén disponibles, así como introducir un título al trabajo (3) Permite aportar información sobre el sitio de unión (4) Permite introducir restricciones sobre la distancia mínima y máxima a la que interaccionan las macromoléculas.
- En nuestro caso el código PDB de la interacción entre MDM2-p53 es 1YCR (https://www.rcsb.org/structure/1ycr). No necesitamos descargar el archivo pdb. Vamos a introducir simplemente la PDB ID: ChainiD. En “Imput Receptor Molecule” pondremos “1YCR:A” y en “Imput Ligand Molecule” pondremos “1YCR:B”. La cadena A corresponde con el péptido de la proteína MDM2 que interacciona y la cadena B con el péptido de p53, esta información la podemos sacar fácilmente de la descripción que nos aparece en (https://www.rcsb.org/structure/1ycr) bajo el apartado de “Macromolecules”. En este caso consideramos MDM2 como la proteína receptora al ser la que presenta estructuralmente el bolsillo hidrofóbico donde interacciona p53.

- Ya podemos realizar el Docking, dándole a “Submit”. No es inmediato, el servidor tardará unos minutos en completar la tarea. Opcionalmente, podemos introducir nuestro correo, el título del docking y definir parámetros de interacción. Pero no es necesario para que el servidor pueda realizar el docking. Es cierto, que podríamos definir la interacción, pero en este caso vamos a poner a prueba el programa y ver cómo de buena es su capacidad predictiva para detectar el sitio de unión.
- Pasaremos a analizar los resultados. Nos deberá de aparecer una página como la que se muestra en la imagen inferior. (http://hdock.phys.hust.edu.cn/data/60992fde4a4dc/)
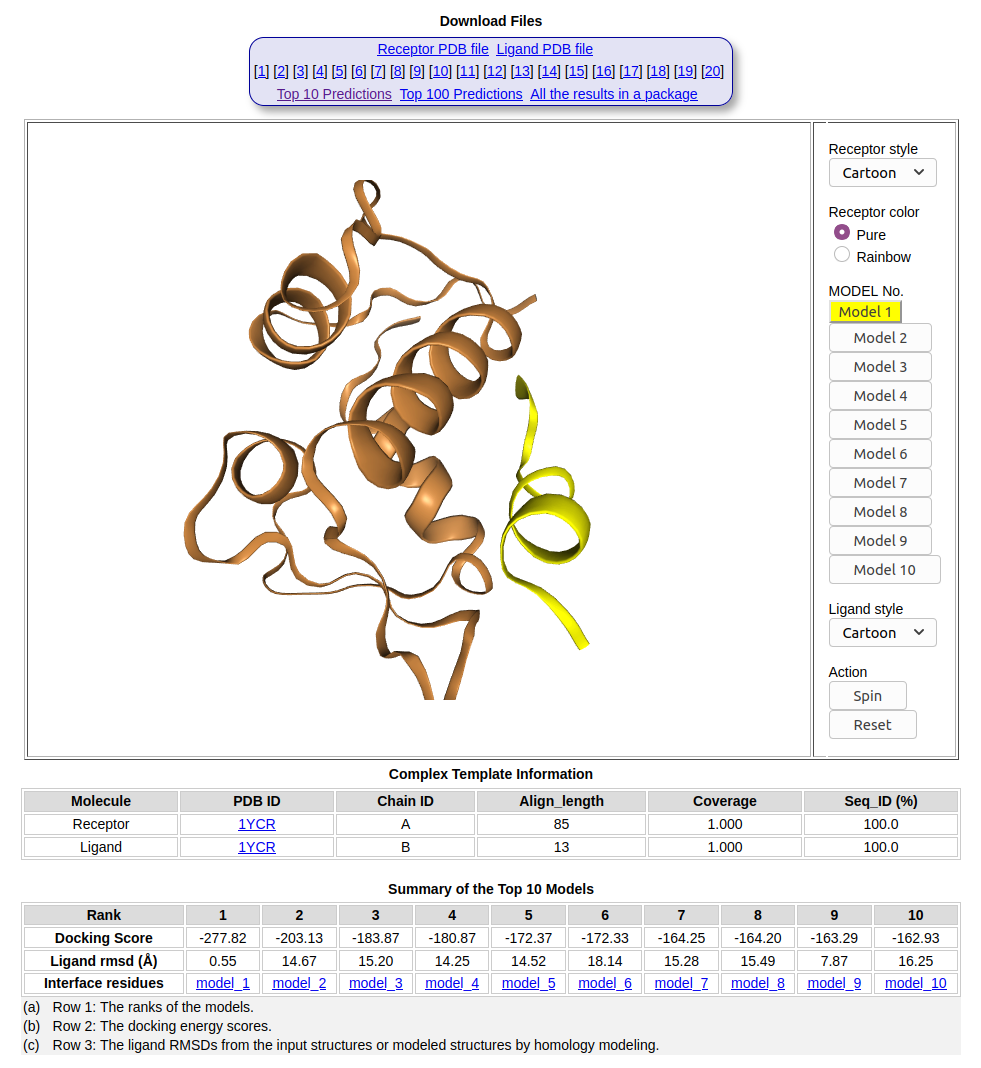
En la parte superior “Download Files” nos aparecen las distintas estructuras pdb que podemos descargar. En la parte derecha, podemos jugar con el estilo en el que queremos que se visualice tanto el receptor como el ligando así como la conformación del ligando. Debajo de la imagen nos aparece una tabla con información del ligando y el Receptor. Recordad que en nuestro caso el ligando es p53 (en la imagen aparece en amarillo) y el receptor es MDM2 (en la imagen aparece en dorado).
Al final de la página nos aparece un resumen de las 10 conformaciones con el “Docking Score” más bajo predecidas por el algoritmo de HDOCK. Cuanto menor sea esta puntuación, más probable estima el algoritmo que sea cierta esa conformación. Nos aparece también información sobre la diferencia entre la estructura predicha y la proporcionada (RMSD) y así como los residuos que establecen interacción.
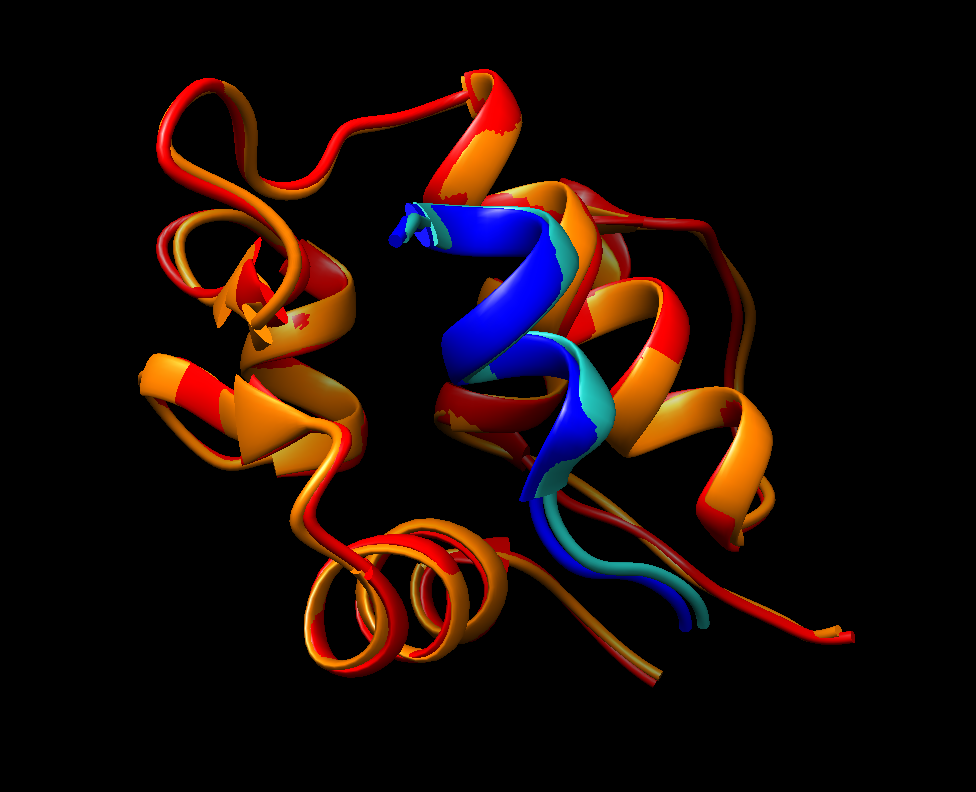
- Vamos a descargar la conformación con menor “Docking Score” y RMSD, dándole a [1] debajo de “Download Files”. Se nos guardará un archivo .pdb de la estructura predecida, que podremos abrir en UCSF Chimera. Este visualizador de macromoléculas como se ha visto en entradas posteriores, presenta multitud de opciones a la hora de visualizar estructuras. Se os deja a vuestra imaginación cómo queréis visualizar la proteína. En nuestro caso, vamos a visualizar la estructura predecida super-poniéndola con la estructura descrita en 1YCR.pdb, descargado de (https://www.rcsb.org/structure/1ycr). Coloreando en naranja la proteína MDM2 del docking y en azul claro p53 del docking. En rojo y azul oscuro aparecen las estructuras descritas obtenidas por difracción de rayos X.
Conclusión
Hoy hemos aprendido a realizar docking macromolecular entre dos proteínas. Por medio de UFC chimera, hemos podido comprobar superponiendo ambas estructuras (la predecida por docking y la descrita) que la predicción estructural es muy buena.
Lo próximo
Se seguirá indagando en el campo de docking por medio de diferentes estrategias y recursos.
Bibliografía
Oliner, JD, Kinzler, KW, Meltzer, PS, George, DL & Vogelstein, B 1992, ‘Amplification of a gene encoding a p53-associated protein in human sarcomas’, Nature, vol. 358, no. 6381, pp. 80–83.
Oliner, JD, Saiki, AY & Caenepeel, S 2016, ‘The Role of MDM2 Amplification and Overexpression in Tumorigenesis’, Cold Spring Harbor Perspectives in Medicine, vol. 6, no. 6, p. a026336.
Yan, Y, Zhang, D, Zhou, P, Li, B & Huang, S-Y 2017, ‘HDOCK: a web server for protein–protein and protein–DNA/RNA docking based on a hybrid strategy’, Nucleic Acids Research, vol. 45, no. W1, pp. W365–W373.