Exploración de los resultados de Swiss Target Prediction
¿Qué vamos a ver hoy?
En esta entrada vamos a llevar a cabo una aproximación de docking macromolecular para comprobar la interacción entre nuestro ligando menadiona y la proteína Cdc25B. Se ha de comprobar, buscando en el portal PDB (https://www.rcsb.org) si se tiene recogida la estructura molecular de esta proteína, buscando bien sea su nombre común o su ID Uniprot.
El docking se va a llevar a cabo con el servidor web “SwissDock”, cuya finalidad y uso se ha comentado en otras entradas de blog: REALIZACIÓN DE UN DOCKING EN SWISS-DOCK
Para entender por qué se está usando SwissDock primero se tiene que definir dos tipos de docking:
- “Blind Docking”: En esta modalidad de docking no se definen los sitios de unión de la proteína, sino que que se realiza el docking con la proteína entera. Se lleva a cabo en casos en los que no se conozca el sitio catalítico/ sitio de unión de la proteína con el ligando. Ejemplo: SwissDock
- “Site Specific Docking”: En esta modalidad de docking sí se definen los sitios de unión de la proteína, se definen qué residuos se conoce que interaccionan con el ligando. Ejemplo: Autodock
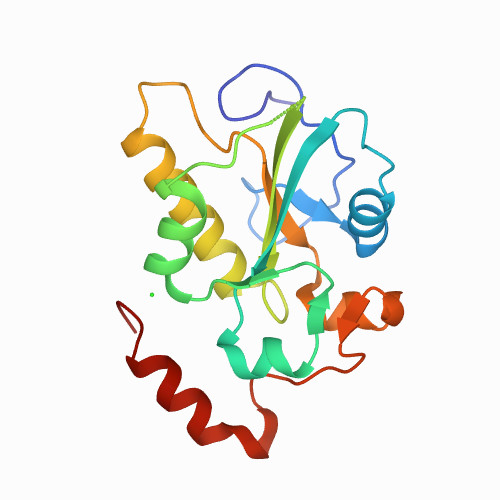
En este caso, a pesar de que se haya descrito interacción entre la proteína Cdc25B y el ligando menadiona no se ha determinado el sitio de unión. Por tanto se ha de llevar a cabo “Blind Docking” para lo que SwissDock es ideal. Es un programa muy sencillo usado desde la página web sin embargo hay limitaciones que se han de considerar. El tamaño de la proteína “target” está limitado a un máximo de 150.000 átomos. Es por ello que para la proteína Cdc25B se va a seleccionar un dominio nada más y no la proteína entera. Se ha empleado la estructura con código PDB “1YMK” que corresponde con la estructura del dominio catalítico de CDC25B.
Para la selección del “target” se puede bien buscar por código PDB, nombre de proteína, secuencia o URL; o bien subir un archivo en formato PDB o charmm. En nuestro caso, subiremos el archivo pdb de nuestra proteína “Dual specificity phosphatase Cdc25B ”. Para la selección del ligando podemos buscar el número de accesión de ZINC (base de datos donde se recogen diferentes estructuras de moléculas), el nombre del ligando o URL; o bien subir un archivo en formato mol2 o charmm. En nuestro caso introduciremos el número de accesión de la menadiona en la base de datos ZINC “ZINC000000001677” (para encontrar su número de accesión basta con realizar una búsqueda de “menadione” en ZINC – http://zinc.docking.org/substances/ZINC000000001677/).
Como resultado nos muestran las diferentes conformaciones que se predicen junto con la energía libre de Gibbs estimada (kcal/mol). Nuestro ligando aparece representada con el esqueleto carbonado en gris, los átomos de oxígeno en rojo y los hidrógenos en blanco.
Aquí os dejamos los enlaces con los resultados de docking (caduca 12-05-2021):
http://www.swissdock.ch/docking/view/swissdockd_xmsTLT_KCNJN9DXC7RWQA3XIF76
Conclusión
Se han buscado targets de la menadiona empleando la herramienta “SwissTargetPrediction” y se ha dado con la proteína Cdc25B – una proteína que controla el ciclo celular y cuya inhibición está asociada con la disminución del desarrollo del cáncer. En base a la literatura, se ha descrito que la menadiona actúa como inhibidor y en base a los resultados de docking, se ha predicho el posible sitio de unión de la menadiona con Cdc25B.